Proteomica e Metabolomica
La Facility di Proteomica e Metabolomica fornisce prestazioni analitiche ad alta sensibilità (inclusi metallomica e lipidomica) utilizzando strumenti e metodi di spettrometria di massa all'avanguardia.
La missione della struttura è di fornire un ambiente di lavoro stimolante che garantisca la gestione di strumenti di spettrometria di massa e lo sviluppo di competenze che implementino ed affianchino le normali risorse disponibili nell’ambito dei singoli progetti di ricerca. Inoltre, l’Unità mira a fornire un ambiente ricco di competenze e risorse formative, a sviluppare e validare nuove metodiche di ricerca e infine, a promuovere la collaborazione sia all'interno della comunità scientifica UdA che all’esterno.
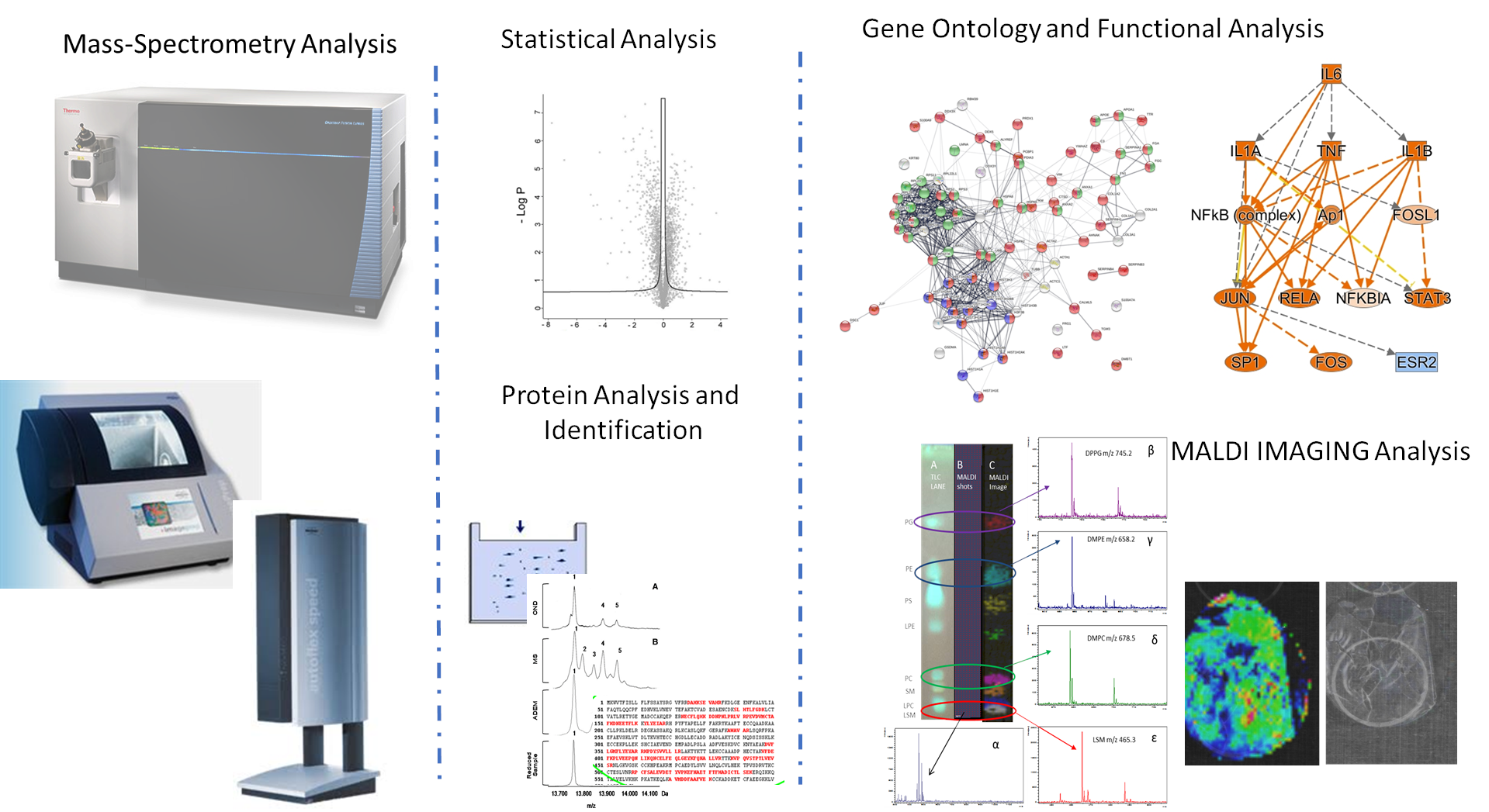
Applicazioni di proteomica
Gli approcci in grado di chiarire l'identità delle proteine, le loro funzioni e interazioni in sistemi cellulari, fluidi biologici, mezzi condizionati, strutture subcellulari o tessuti, includono:
- LC-MS/MS per l’Identificazione delle proteine da campioni digeriti ottenuti da banda/spot su gel, frazioni cromatografiche, frazioni immunoprecipitate o campioni complessi (fluidi biologici, tessuti, esosomi arricchiti e vescicole extracellulari)
- Quantificazione relativa del proteoma mediante approcci label-free, utilizzando sistemi LC-MS e LC-MS/MS ad alta risoluzione ed accuratezza di massa
- LC-MS/MS per l’identificazione delle modifiche post-traslazionali delle proteine (PTM)
- Analisi LC-MS di Proteomica top-down ad alta risoluzione direttamente su proteine intatte
- MALDI-MS (MS/MS) per analisi di profili proteici e peptidici
- MALDI-TOF-MS IMAGING per l’analisi proteomica su campioni tissutali
- Approcci di metallomica quantitativa in diverse matrici biologiche (urina, plasma, terreni per colture cellulari, cibo, bevande) per diversi elementi quantificabili (Arsenico, Berillio, Cadmio, Cobalto, Cromo, Ferro, Iodio, Manganese, Nichel, Piombo, Platino, Rame, Zinco)
Applicazioni di metabolomica
I nostri metodi avanzati di metabolomica consentono di rivelare e quantificare diverse classi di molecole in una varietà di matrici biologiche, in modalità targeted ed untargeted. Le tecnologie disponibili includono:
- Metodi quantitativi specifici per l'analisi di metaboliti polari, steroidi, farmaci e lipidi
- Metodi di analisi untargeted attraverso spettrometria di massa ad alta risoluzione LC-MS e LC-MS/MS per la quantificazione relativa di metaboliti polari e per il profiling di lipidomica
- MALDI-TLC-IMAGING di lipidi polari
- MALDI-TOF- MS-IMAGING di lipidi polari
INFORMAZIONI PER L’ACCESSO
Questa piattaforma condivisa fornisce analisi ad alta sensibilità utilizzando strumenti e metodi di spettrometria di massa all'avanguardia. A causa dell'elevata specificità e complessità delle strumentazioni, per ogni applicazione è richiesta la collaborazione del personale specializzato della struttura provvisto di competenze necessarie per gestire la sofisticata strumentazione disponibile e svilupparne le applicazioni.
Spettrometro di Massa “Thermo Scientific Orbitrap Fusion Tribrid”
dotato di:
- U3000 NANO RSLCnano SYS for Easy-Spray
- UltiMate 3000 RSLCnano for Easy-Spray
MALDI TOF/TOF autoflex speed Bruker Daltonics
dotato di:
- Dispositivo di sistema ImagePrep per configurazione IMAGING (Bruker Daltonics)
- GC-MS – 6890N GC con un autocampionatore serie 7863, accoppiato a spettrometro di massa 5973N (Agilent Tecnologies).
- ICP-MS – spettrometro di massa quadrupolare ICP 7500 (Agilent Technologies)
- HPLC-ESI Triple Quadrupole MS (TQUATTRO Ultima Pt, Micromass-WATERS)
Software
- MaxQuant (Istituto Max Planck di Biochimica, Martinsried, Germania)
- Perseus (strumenti statistici per l'analisi dei dati “omici”)
- Ingenuity Pathways Analysis “IPA” (integrazione e interpretazione funzionale dei dati derivati da esperimenti “omici”)
- MetaboAnalyst (analisi statistica, funzionale e integrativa dei dati di metabolomica)
 Prof. Piero Del Boccio, PhDHead of Proteomics and |
||
 Dr. Ilaria Cicalini, PhDShotgun proteomics; Bioinformatics; |
 Dr. Ada Giovanna Consalvo, PhDTargeted metabolomics; |
